Las tijeras moleculares CRISPR-Cas9 reciben el Nobel de Química
Miércoles, 7 de Octubre de 2020. Lo primero que veo al despertar es una oleada de notificaciones en mi teléfono, y no es para menos. Hace pocas horas se había anunciado a Jennifer Doudna y Emmanuelle Charpentier como galardonadas con el premio Nobel de Química “por el desarrollo de un método de edición genética”, comúnmente conocido como CRISPR-Cas (Imagen 1). Al formar parte activa de este campo no pude dejar de emocionarme y correr hacia mi laboratorio para ver las reacciones de mi jefe y compañeros. Mi jefe, Dr. Mitch O’Connell, quien previamente había trabajado en el laboratorio de la doctora Doudna antes de montar su propio laboratorio, ya nos había dejado una pequeña sorpresa al llegar (Imagen 2).


Pero, ¿qué es CRISPR-Cas9 y por qué tanta importancia? ¿Por qué oímos tanto sobre “tijeras moleculares”? El objetivo de esta entrada será explicar muy brevemente qué es CRISPR, la historia de su descubrimiento y cómo se logró desarrollar en una herramienta de edición genética.
Para empezar a entender CRISPR, debemos saber que no somos los únicos organismos que nos vemos afectados por virus. Prácticamente cada organismo que existe en la Tierra es susceptible de ser atacado por algún tipo de virus, y los organismos procariotas (bacterias y arqueas) no son la excepción. Estos virus reciben el nombre de bacteriófagos o simplemente fagos. En general, sucumbir ante el virus supone la destrucción del huésped en el proceso. Al considerar que los fagos son la entidad biológica más abundante del planeta (~10,000,000,000,000,000,000,000,000,000,000 o ~1031) y que exceden 10 veces el número de procariotas, no es difícil imaginar que una interminable guerra biológica se desarrolla cada segundo a escala microscópica (Imagen 3).
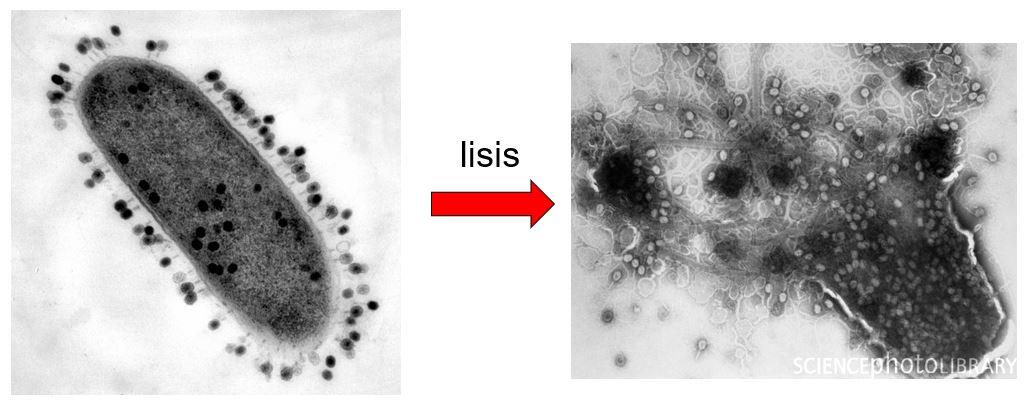
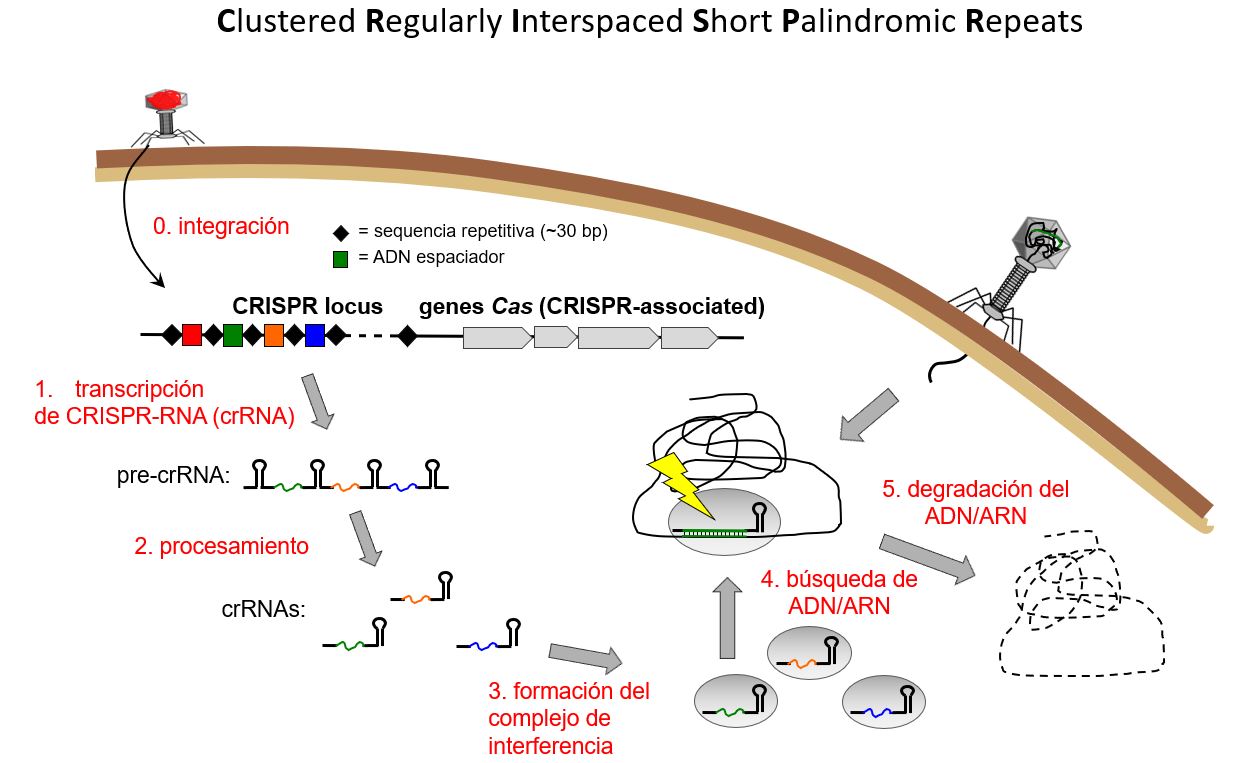
A pesar del sinnúmero de estrategias moleculares que presentan estos virus, los procariotas no están del todo indefensos ante estos ataques indiscriminados. Millones de años de evolución les han permitido desarrollar defensas ante estos agresores. Y aunque muchas de estas defensas son innatas, existen algunas defensas mucho más específicas conocidas como adquiridas. Este es el caso del sistema bacteriano CRISPR. CRISPR, acrónimo del inglés Clustered Regularly Interspaced Short Palindromic Repeats hace referencia a una serie de secuencias repetitivas en el ADN de muchas especies de bacterias o arqueas capaces de conferir protección y adaptación contra virus y elementos extraños.Las bacterias, mediante este sistema inmune adaptativo y tras tener un encuentro con alguno de estos virus, cortan el material genético de estos invasores y partes de este material son incorporadas al propio ADN bacteriano. El lugar donde se colocan estos fragmentos es entre estas secuencias repetitivas. Es como una memoria o un póster indicando: “Se Busca, virus peligroso”, lo que permite a la bacteria reconocer rápidamente y destruir a invasores similares (para los más curiosos, referirse a la Imagen 4 adjunta con más detalle). ¿Cómo? Al encontrar material genético similar al almacenado en esta “base de datos”, se generan copias de esta información que dirigen a toda una maquinaria para destruir el material genético del virus, anulando su capacidad infectiva y resultando en protección para la bacteria o arquea.
La existencia de CRISPR fue un descubrimiento tardío. Alrededor de 1993, el investigador alicantino Francisco Mojica – en aquel entonces estudiante de doctorado en la Universidad de Alicante – fue el primero en observar un patrón repetitivo inusual en el ADN de algunas especies que viven en las aguas salinas de Santa Pola. Al indagar más a fondo descubrió un artículo de un grupo japonés de 1989 que había visto algo similar en otra especie de bacteria (E. coli), aunque para ellos no fue más que una observación trivial. Pero esto despertó una gran curiosidad en Mojica, quien se dispuso a buscar fenómenos similares en otras especies y publicó sus hallazgos iniciales en 1995. Tras volver como profesor a la Universidad de Alicante tras una estancia posdoctoral en Oxford, con fondos y recursos mínimos, decidió revisitar este proyecto usando bioinformática. De esta forma encontró muchas más especies que poseían estas extrañas repeticiones en su ADN que fueron bautizadas por él mismo como CRISPR. Las hipótesis sobre la función natural de las repeticiones CRISPR no se hicieron esperar: regulación de genes, reparación del material genético, etc. Ninguna de ellas correctas y fue el mismo Mojica quien, al examinar la secuencia de este material genético – cada una de las 4 letras químicas que se combinan para dar un mensaje en el ADN – descubrió rápidamente que un buen número de ellas no tenían nada que ver con información genética de esa especie, sino que era información de virus que las atacaba. En un giro de los acontecimientos, Mojica entendió entonces que CRISPR codifica instrucciones para un sistema adaptativo inmune que protege a estos microbios contra infecciones específicas. A pesar de estas emocionantes noticias, ninguna revista de alto prestigio consideró este trabajo meritorio de publicación y el investigador alicantino tuvo que navegar a través de diferentes revistas antes de que pudiera ser publicado. Pero finalmente, con estos trabajos, Mojica puso en acción una serie de acontecimientos que llevaron no solo a avanzar el campo de la microbiología, si no en su momento, a la edición genómica.

La presencia de CRISPR fue un hallazgo emocionante entre los microbiólogos y otros no tardaron en identificar más y más especies que poseían CRISPR. Muy posiblemente, la falta de recursos de Mojica impidió que pudiera demostrar de forma fehaciente esta función protectora, pero finalmente Philip Horvath lo hizo, amparado por la industria del yogur. La fermentación del yogur se realiza a través de ciertas especies de bacterias por lo que las contaminaciones con bacteriófagos en una planta de producción son desastrosas para la industria. Así que no es de extrañar que tuvieran un especial interés en aprender más sobre cómo proteger estas especies fermentadoras de infecciones víricas. Horvath demostró de forma experimental que las secuencias de CRISPR ofrecen protección e incluso demostró que incorporando nuevas secuencias de forma artificial se podía conferir protección adicional contra otros virus.
A partir de entonces la investigación se centró en entender cada uno de los procesos implicados en esta inmunidad. El trabajo de muchos como John van der Oost, Eugene Kooning, Luciano Marraffini, Erik Sontheimer, Jörg Vogel, Virginijus Siksnys y otros fue crucial para diseccionar cada uno de los componentes y procesos implicados en CRISPR. Mientras esto ocurría, en 2011 la investigadora francesa Emmanuelle Charpentier descubrió que un fragmento de ARN (tracrRNA) era imprescindible para dirigir la proteína Cas9 junto con su guía hacía la región invasora que debía ser eliminada. Al mismo tiempo inició una fructífera colaboración con la bioquímica Jennifer Doudna de la Universidad de California Berkeley. Juntas lograron recrear estas tijeras moleculares en un tubo de ensayo, comprendieron mejor su funcionamiento, simplificaron los componentes de este sistema y finalmente, describieron como reprogramar estas tijeras para no solo cortar ADN viral sino para poder cortar cualquier ADN, a voluntad y en sitios específicos, y sustituirlo por otra secuencia deseada. Y entonces todo cambió, porque donde el ADN se corta, se puede reescribir el código de la vida.
Aquí fue cuando dos ramas de la biología se juntaron para dar lugar a uno de los avances más relevantes de las últimas décadas. Allá por 1980, brillantes científicos como Mario Cappecci (premio Nobel en 2007 en fisiología) describieron técnicas para modificar los genomas, es decir, toda la información genética de un individuo contenido en su ADN. El mayor problema era la gran ineficiencia de estos procesos. Pronto, se observó que una rotura del ADN incrementa las posibilidades de poder introducir cambios en la región donde ocurre ese daño. Esto es así porque al haber una rotura en el ADN, la célula activa todas sus alarmas y envía toda una maquinaria para reparar la situación. De lo contrario, la rotura podría ser fatal. Muchas veces la célula solo puede unir los extremos rotos perdiendo algo de información genética lo cual podría ser devastador dependiendo de donde ocurra. Pero si existe ADN similar intacto en las proximidades, la célula utiliza este como molde para copiar este mensaje en la rotura y cerrarla, con la estrategia de que posiblemente era el mismo mensaje contenido inicialmente. Muchos fueron los esfuerzos por poder generar roturas en el ADN y enviar moldes de ADN que facilitaran modificaciones a voluntad, pero llevaba mucho tiempo, dinero, esfuerzo y muchas veces terminaba en fracaso (para más información, recomiendo leer sobre dedos de Zinc y TALENS).
Al presentar un método basado en CRISPR fácil de usar, relativamente barato, eficiente y totalmente programable, Doudna y Charpentier ofrecieron al mundo una tecnología que permitiría modificar la información genética de un individuo a voluntad. Y así, un sistema inmune bacteriano ancestral se convirtió en una herramienta de edición genómica (Imagen 6).
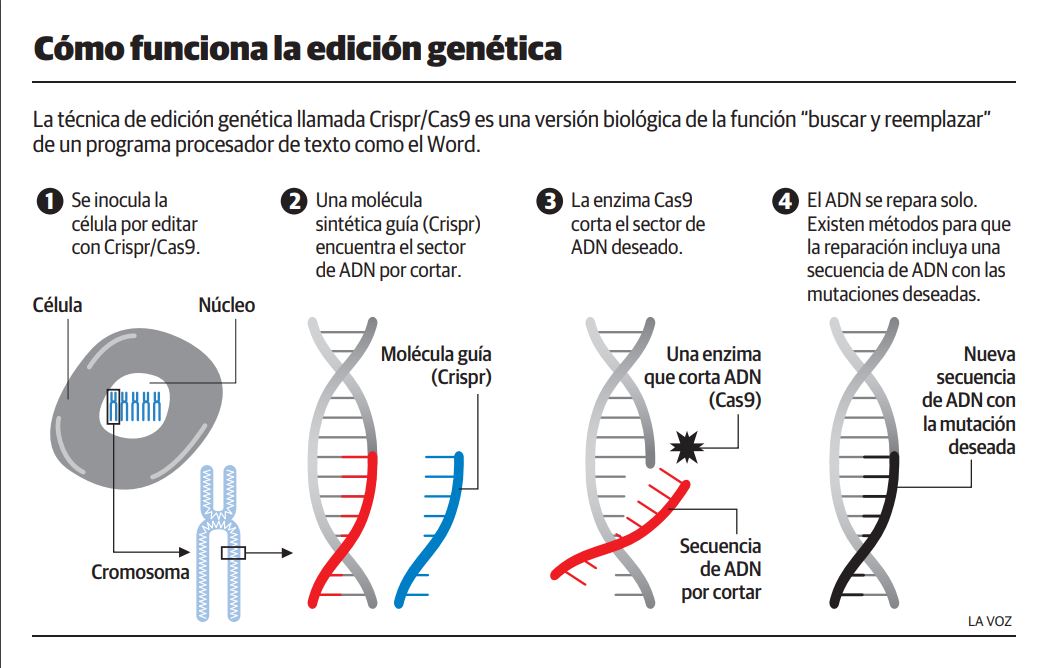
Naturalmente requirió algo más de trabajo y esfuerzo poner a punto CRISPR-Cas9 (destacamos el trabajo de Feng Zhang y George Church entre otros pioneros) pero hoy en día prácticamente la inmensa mayoría de laboratorios de investigación biomédica y biológica utilizan sistemas CRISPR como herramienta. Se han podido generar modelos celulares y animales de enfermedades humanas que hasta ahora no habían sido fáciles de generar, se está utilizando en plantas y ya hay numerosos esfuerzos para curar enfermedades genéticas humanas. Las posibilidades son ilimitadas.
Aunque su uso en investigación es abundante, la tecnología aún no está preparada para modificar de forma permanente el genoma de un ser humano. En primer lugar, las regulaciones bioéticas prohíben expresamente este tipo de práctica y, en segundo lugar, la seguridad de usar esta tecnología no está garantizada al 100%. Se sabe que en ocasiones Cas9 puede cortar en regiones similares a la deseada pero que no es la diana planeada.
Lo que es seguro es que llegará el día en el que todos tomemos una decisión sobre cuál será el futuro de las modificaciones genéticas y debemos educarnos y estar preparados para tomar una decisión informada. Aunque pudiéramos, ¿deberíamos tener el poder de modificar la información genética de la especie humana o de cualquier especie? Por ahora, te dejo que lo medites. Lo que es seguro es que un descubrimiento en las salinas de Alicante, basado en investigación básica y, en apariencia, insignificante, ha llevado a una de las mayores revoluciones en Ciencias de la Vida de este siglo. Quizá esta experiencia nos sirva de lección para valorar y financiar mejor la investigación básica. Todo conocimiento nuevo es precioso y no sabemos a dónde nos llevará.
Glosario
Arquea: Uno de los tipos de procariotas (organismos unicelulares sin núcleo ni orgánulos membranosos) pero que se diferencian del otro grupo (bacterias) en muchos aspectos de su genética y bioquímica, y que muestran una historia evolutiva independiente. Son comúnmente frecuentes en ambientes hostiles, donde otros organismos no podrían desarrollarse (altas temperaturas, salinidad, etc.).
Defensa innata: Hace referencia a los mecanismos inmunitarios que actúan de forma genérica y no específica contra un patógeno. Es una respuesta rápida pero no deja memoria.
Defensa adquirida: Componen sistemas especializados encaminados a reconocer y recordar patógenos para su eliminación. Aunque su actuación es más lenta, son sistemas altamente específicos y dejan memoria para defenderse ante futuros encuentros con dicho patógeno.
Locus (plural, loci): Nombre que hace referencia al lugar concreto en el ADN donde reside un determinado gen o un marcador genético.
E. coli: Especie de bacteria muy común en los intestinos de personas y animales, aunque algunas cepas pueden causar enfermedades intestinales. Se ha utilizado como modelo procariota para descubrir muchos procesos en biología molecular y actualmente es utilizada como herramienta en microbiología, biotecnología y especialmente útil en técnicas de ADN recombinante.
Bioinformática: Subdisciplina de la biología e informática, que se concentra en la adquisición, almacenamiento, análisis y tratamiento de datos biológicos, sobre todo secuencias de ADN y de aminoácidos.
Bioética: Rama de la ética dedicada a promover los principios para la conducta más apropiada del ser humano con respecto a la vida.
Para saber más
- Aplicaciones del CRISPR: 1 y 2.
- Noticia sobre los trabajos de Doudna y Charpentier, galardonados con el Nobel de Quimica del 2020.
- Desarrollo de sistemas CRISPR para detectar SARS-CoV2.
- CRISPR explicado por el mismo Mojica: 1 y 2.
- Genome Editing with CRISPR-Cas9 (en inglés).
Sobre el autor: Adrian Molina Vargas
Lo mejor que se puede hacer en la vida es viajar, y es lo que más me gusta.
También me encanta leer y disfrutar de la comida en buena compañía.
Me gradué en Biología Sanitaria (UAH) y actualmente estoy realizando el doctorado en Genética (University of Rochester), investigando los recién descubiertos sistemas CRISPR-Cas13, para generar herramientas eficaces y específicas para detectar y manipular el ARN. Posiblemente sea el más raro cuando diga que mi bebida favorita es la sidra. #HardCider

